
Contador de células
En el contador de células: Célula HepG2
HepG2 es una línea celular obtenida a partir del tejido hepático de un paciente con carcinoma hepatocelular (CHC). A menudo se utiliza como sistema modelo del CHC, así como para estudios de metabolismo y toxicidad de fármacos. Los cultivos son adherentes, con morfología epitelial, y tienden a crecer en pequeños agregados que dificultan el recuento de células individuales. En las condiciones de cultivo adecuadas, pueden formar superficies apical y basal diferenciadas aptas para estudios de enfermedades hepáticas.
Ver el seminario web de detección de células StainFree
Descargar la nota de aplicación de detección de células StainFree
Descargar la nota de aplicación de detección de células StainFree (China)
Descargar eBook: Realice recuentos de células de forma profesional
Figura 1: Determinación mediante StainFree del porcentaje de área cubierta

Se adquirieron imágenes de las células HepG2 con el citómetro con adquisición de imágenes SpectraMax MiniMax 300 y las células se identificaron utilizando una configuración de análisis personalizada, definida por el usuario. A la izquierda se muestran las imágenes originales con luz transmitida y a la derecha, la misma imagen con máscaras púrpura que indican las células identificadas por el software. Se muestran dos densidades de células para su comparación.
Figura 2: Análisis StainFree frente a fluorescente del porcentaje de área cubierta
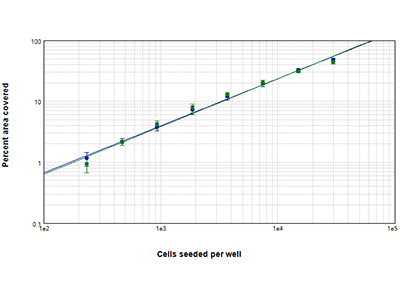
La confluencia de las células HepG2 sembradas a densidades que oscilaban de 234 a 30 000 células por pocillo se analizó usando la tecnología StainFree (puntos azules) o se tiñeron con el colorante de ensayo EarlyTox™ Live Cell y se analizó el área fluorescente (puntos verdes). El porcentaje de área cubierta en una región de interés de la imagen para ambos métodos coincidía bastante en el rango completo de densidades de células.
Figura 3: Ensayo EarlyTox Caspase-3/7 NucView 488
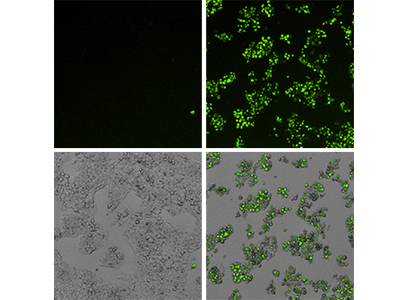
Las células HepG2 tratadas con medio de control (paneles izquierdos) o capsaicina a 200 µM (paneles derechos) se analizaron con el kit de ensayo EarlyTox Caspase-3/7 NucView 488 y las imágenes se obtuvieron utilizando el citómetro con adquisición de imágenes SpectraMax MiniMax 300 con los canales de fluorescencia verde y de luz transmitida (LT). Fila superior: imagen de fluorescencia que muestra las células apoptóticas marcadas con fluorescencia verde. Fila inferior: imágenes de LT y fluorescencia superpuestas que muestran la apoptosis mínima en las células control y cerca del 100 % de apoptosis en las células tratadas.
Figura 4: Ensayo EarlyTox Caspase-3/7 NucView 488 (evolución temporal)
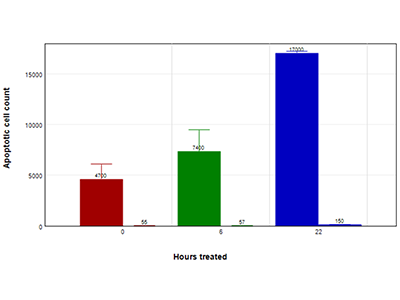
Células HepG2 tratadas con 200 µM de capsaicina (barra izquierda) o medio de control (barra derecha) durante 4,5 horas (barras rojas), 6 horas (barras verdes) o 22 horas (barras azules). La apoptosis se evaluó usando el kit de ensayo EarlyTox Caspase-3/7 NucView 488. Las células se incubaron con 5 µM de sustrato NucView 488 y se adquirieron imágenes en los puntos temporales indicados.
Figura 5: Kit de ensayo EarlyTox Caspase-3/7 R110 y kit de ensayo EarlyTox Glutathione
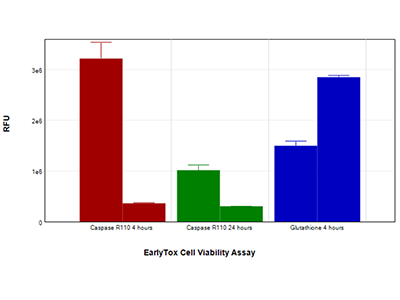
Las células HepG2 tratadas con 200 µM de capsaicina (barra izquierda de cada par) o medio de control (barra derecha de cada par) se analizaron con el kit de ensayo EarlyTox Caspase-3/7 R110 (barras rojas y verdes), que mide la actividad caspasa, o con el kit de ensayo EarlyTox Glutathione (barras azules), que mide la disminución del glutatión, un indicador temprano de apoptosis. Ambos ensayos se detectaron en el lector de microplacas multimodo SpectraMax i3x utilizando protocolos preconfigurados del software SoftMax Pro.
Consejo: Las células HepG2 crecen en agregados de células con forma irregular que son indistinguibles a simple vista. Una forma útil de cuantificar el crecimiento o la confluencia de las células HepG2 es mediante el uso de tecnología StainFree™ para determinar el área cubierta por las células. Las imágenes obtenidas en el canal de LT se pueden analizar fácilmente usando la configuración “Field Analysis” (Análisis de campo). Se puede utilizar la configuración de análisis “Cells” (Células) predefinida o se puede diseñar un análisis personalizado utilizando las herramientas de dibujo del software SoftMax Pro.
Kit de herramientas de análisis de células HepG2
- Plataforma de detección de microplacas multimodo SpectraMax® i3
- Citómetro con adquisición de imágenes SpectraMax® MiniMax™ 300
- Software SoftMax® Pro
Configuración del instrumento
Tipo de análisis: Field Analysis (Análisis de campo)
Longitud de onda para la búsqueda de objetos: LT
Acerca de la tecnología de detección de células StainFree
Los ensayos basados en la adquisición de imágenes celulares normalmente requieren el uso de sondas fluorescentes que pueden ser tóxicas o funcionar únicamente en células fijadas. Un método sin marcaje para analizar recuentos celulares y la confluencia celular permite a los investigadores monitorizar cuantitativamente la proliferación celular y la salud de las células sin necesidad de flujos de trabajo laborioso que pueden alterar la viabilidad celular.
La plataforma de microplacas multimodo SpectraMax i3 con el citómetro con adquisición de imágenes MiniMax 300 utiliza la exclusiva tecnología de detección de células StainFree pendiente de patente que le permite realizar ensayos de proliferación celular, citotoxicidad y otros ensayos sin tinciones nucleares como DAPI, que se intercala en el ADN, o colorantes de células vivas que son realmente tóxicos para las células a largo plazo.