
Software de automatización de microscopía y análisis de imágenes MetaMorph [Descatalogado]
*LEER* Aviso de retirada del mercado
Molecular Devices evalúa continuamente las tecnologías de análisis de imágenes y automatización de microscopía para garantizar que un producto cumple con las expectativas de nuestros clientes. Tras una cuidadosa consideración, hemos decidido dejar de ofrecer el paquete de software MetaMorph, lo que incluye MetaFluor y MetaVue, después del 7 de julio de 2023. Esta decisión se ajusta a nuestra estrategia de proporcionar nuevos productos innovadores con seguridad, diseño y funcionalidad mejorados en apoyo de la investigación científica.
Software de adquisición de imágenes de microscopio con software de análisis de microscopía opcional
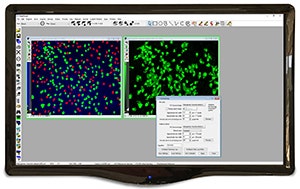
El software de automatización de microscopía y análisis de imágenes MetaMorph® automatiza la adquisición, el control del dispositivo y el análisis de imágenes. Integra fácilmente hardware y periféricos de microscopios de fluorescencia diferentes en una única estación de trabajo personalizada. El software ofrece muchos módulos de aplicaciones fáciles de usar para análisis específicos de biología. En la gama se incluyen dos paquetes complementarios, el software MetaFluor® para la adquisición de imágenes ratiométricas de fluorescencia y el software MetaVue® para la adquisición y procesamiento de imágenes básicas.

Compatible con microscopios de otros fabricantes
El software MetaMorph funciona con muchos microscopios, lanzamientos de láser y óptica TIRF disponibles en el mercado, y se puede habilitar en sistemas de adquisición de imágenes previamente instalados compatibles con el software MetaMorph.

Mida las imágenes ratiométricas de fluorescencia
El software de adquisición de imágenes ratiométricas de fluorescencia MetaFluor® está diseñado para mediciones de iones intracelulares de longitud de onda única o múltiple para ofrecer más datos acerca del intercambio de iones y la función intracelular.

Documente y analice las imágenes
El sistema de adquisición de imágenes de investigación MetaVue™ es una aplicación de software sencilla y fácil de usar para adquirir y procesar imágenes, realizar funciones gráficas y archivar y recuperar imágenes.
Características

Procesamiento de imágenes en tiempo real
El procesamiento de imágenes es asistido por una aceleración por hardware de la unidad de procesamiento gráfico. Resuelve objetos subcelulares de tan solo 20 nm espacialmente y 40 nm axialmente.

Módulo de adquisición multidimensional
Permite la captura de secuencias de adquisición complejas con una interfaz de usuario guiada flexible.
Análisis morfométrico integrado
Mide y clasifica objetos en clases discretas definibles por el usuario en función de cualquier combinación de parámetros morfométricos, como la forma, el tamaño o la densidad óptica.

Módulo de barrido de cortes
Automatiza la adquisición de múltiples imágenes y luego las une perfectamente. Ideal para muestras grandes de tejido, esto asegura la reproducibilidad al tiempo que elimina las suposiciones en los experimentos de mosaico.

Transmisión entre dispositivo y cámara
Acelera la velocidad de captura de imágenes y transmite simultáneamente imágenes a la memoria durante la adquisición, capturando eventos celulares dinámicos para aplicaciones como la adquisición de imágenes cinéticas y en vivo.

Visor 4D con mediciones 3D
Las herramientas para visualización multidimensional incluyen stacks de imágenes secuenciales, múltiples secciones Z, longitudes de onda, puntos temporales y posiciones. Los datos se pueden representar para la visualización y rotación de isosuperficies 3D.
Recursos más recientes
Aplicaciones destacadas
Adelantos para los clientes

HISTORIA DE ÉXITO
