Selectores de colonias microbianas QPix
Sistema automático de cribado microbiano capaz de seleccionar hasta 3000 colonias por hora
Sistema automático de cribado microbiano capaz de seleccionar hasta 3000 colonias por hora
El selector de colonias microbianas QPix® aprovecha la mejor tecnología de selección de colonias de su clase para aliviar los cuellos de botella y realizar cribados de forma rápida, exacta y eficiente de bibliotecas genéticas masivas. El software intuitivo y fácil de usar guía a los usuarios a través de la configuración de los ciclos de selección de colonias en los que la robótica de precisión selecciona siempre las colonias correctas. Además del cribado microbiano, el sistema automatiza varios procesos de preparación de muestra y manipulación de placas, tales como transferencia de cultivo líquido bacteriano y siembra en placa o agar.
Los datos se registran automáticamente en la base de datos del aparato, lo que proporciona a los usuarios una pista de auditoría completa y seguimiento de la muestra, garantizando que nunca se pierdan datos. Nuestra serie modular y ampliable de selectores de colonias permite a grupos de cualquier tamaño aumentar la exactitud y el rendimiento de sus flujos de trabajo, permitiendo a la vez incrementar el rendimiento en el futuro.

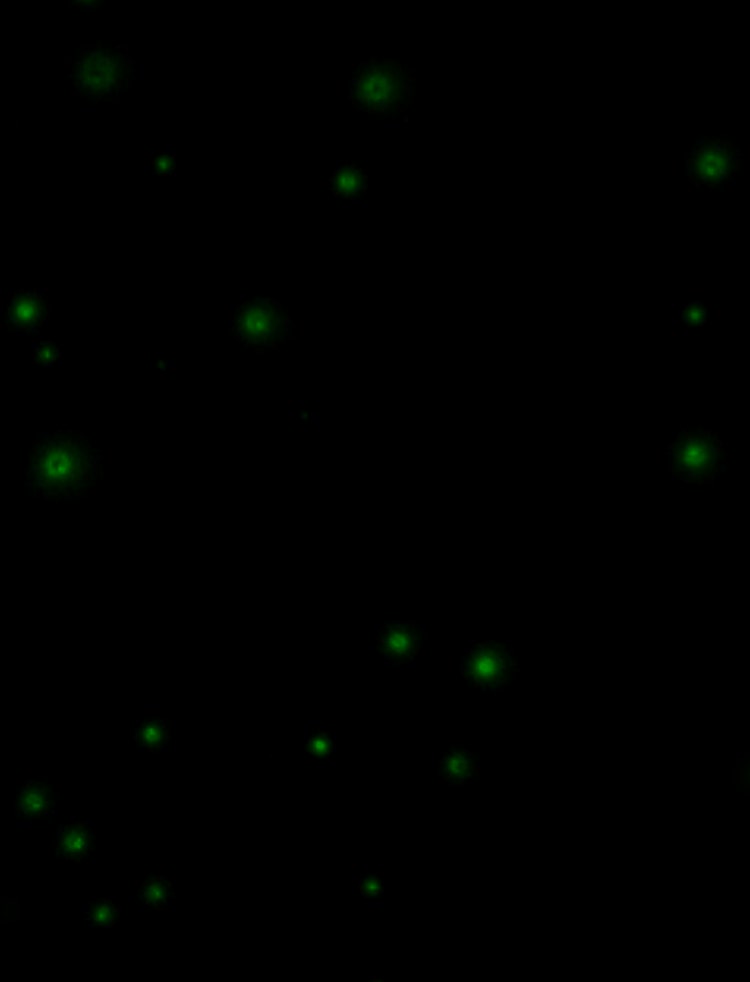
Identifique colonias con el fenotipo deseado
Los selectores de colonias QPix admiten una amplia variedad de microorganismos y diferentes modalidades de selección, como intensidad de fluorescencia, selección azul/blanco, tamaño y proximidad y zona de inhibición.

Seleccione colonias de forma eficiente
Un juego de sonda específica de organismo y sensor de agar garantizan una selección eficiente. El sistema proporciona una selección eficiente >98 %, lo que le permite disponer de tiempo con confianza.

Mantenga la esterilidad
Hay disponible una serie de características de esterilidad, incluido un proceso de luz UV para desinfectar el interior del instrumento, así como el lavado y secado con halógeno de las sondas.
Anatomía de un sistema QPix
Características

Sondas específicas de organismo
Las sondas de diferentes formas y áreas de selección maximizan la eficiencia para E. coli, fagos y levadura. Las sondas específicas de siembra garantizan una distribución uniforme del cultivo líquido sobre el agar.

Modos múltiples de adquisición de imágenes
Las colonias se pueden seleccionar en función de parámetros preespecificados usando luz blanca, fluorescencia y color. El uso de filtros permite aplicaciones como el cribado de colonias blancas/azules.

Siembra y extensión
Se puede realizar la siembra y estriado automático de 96 muestras en 30 minutos, lo que permite disponer de más tiempo.

Replicación, cuadrícula y selección de positivos
La manipulación y el seguimiento automatizados de placas optimizan el ensayo posterior y la gestión de las muestras. Los selectores de colonias QPix proporcionan funciones flexibles de replicación y cuadriculación de placas y selección de colonias positivas.

Detección de agar
El sensor ultrasónico de la altura del agar detecta diferencias en la altura debidas al volumen variable de vertido, lo que permite la máxima eficiencia de la selección.

Opciones de automatización adaptables*
El modelo QPix HT es una solución compatible con robots con una plataforma modular. El equipo de Soluciones Avanzadas de Ingeniería para Flujos de Trabajo puede adaptar un selector de colonias con una serie de servicios personalizados.
* El precio, el plazo de entrega y las especificaciones pueden variar en función de los requisitos técnicos acordados mutuamente. Los requisitos de la solución pueden causar ajustes en el rendimiento estándar.
Automatice su flujo de trabajo con el selector de colonias QPix
La selección de colonias es un paso esencial en la investigación biomédica, ya que los científicos aíslan con frecuencia clones microbianos para producir en masa ADN o proteínas para su uso en diferentes aplicaciones posteriores. Tradicionalmente, la selección de colonias se realiza manualmente, empleando puntas de pipeta o asas de inoculación estériles, lo que supone un proceso lento, laborioso y largo. Los selectores automáticos de colonias no solo harán más rápido el proceso completo, sino que los resultados son más uniformes y fiables.
¿Cuál es el QPix adecuado para usted?
imaging
picking-capacity
colony-selection-criteria
picking-and-regional-picking
barcode-tracking
re-arraying-and-replication
gridding
plating-and-streaking
agar-to-agar
robotics-integration
shaking-incubator
liquid-handler
pcr
sealer-peeler
elisa
destination-plate-capacity
stackers
source-plate-capacity
walkaway-time
* El precio, el plazo de entrega y las especificaciones pueden variar en función de los requisitos técnicos acordados mutuamente. Los requisitos de la solución pueden causar ajustes en el rendimiento estándar.
Las soluciones personalizadas están sujetas a las Condiciones de compra de productos personalizados de Molecular Devices disponibles en https://moleculardevices.com/custom-products-purchase-terms.
Preguntas frecuentes
¿Qué es un selector de colonias microbianas QPix?
El selector de colonias microbianas QPix es un avanzado sistema automatizado diseñado para la selección de alto rendimiento de colonias microbianas. Optimiza el proceso de cribado de clones y elimina la necesidad de seleccionar colonias manualmente.
¿Cómo funciona el selector de colonias microbianas QPix?
El selector de colonias microbianas QPix utiliza una combinación de tecnología de adquisición de imágenes y robótica para identificar y seleccionar de forma precisa colonias microbianas individuales a partir de placas de agar. Utiliza algoritmos sofisticados para analizar el tamaño, la forma y otras características de las colonias, lo que permite una selección precisa de estas.
¿Cuáles son las características clave del selector de colonias microbianas QPix?
El selector de colonias microbianas QPix ofrece varias características clave, como la adquisición de imágenes de alta resolución, criterios personalizables de selección de colonias, selección y redisposición de matrices automatizadas, parámetros de selección ajustables y un software intuitivo para el seguimiento de colonias y el análisis de datos.
¿Cuáles son las ventajas del selector de colonias microbianas QPix?
Gracias a la automatización del proceso de selección de colonias, el selector de colonias microbianas QPix aumenta considerablemente el rendimiento y la eficiencia del cribado de clones. Minimiza el riesgo de contaminación cruzada, mejora la exactitud y la reproducibilidad, y libera tiempo y recursos valiosos para los investigadores.
¿Qué aplicaciones se pueden beneficiar del selector de colonias microbianas QPix?
El selector de colonias microbianas QPix es ideal para una gran variedad de aplicaciones, como expresión de proteínas recombinantes, descubrimiento de anticuerpos, biología sintética, evolución dirigida e ingeniería de cepas. Es adecuado para investigación académica, desarrollo farmacéutico, biotecnología y otros sectores que requieren un cribado de clones eficiente.
¿Cuáles son las principales funciones de los selectores de colonias microbianas QPix?
Los selectores de colonias microbianas QPix ofrecen funciones automatizadas de selección, identificación y seguimiento de colonias. Permiten a los investigadores seleccionar y transferir de forma eficiente colonias microbianas individuales desde las placas de agar, ahorrando tiempo y reduciendo el error humano.
¿En qué flujos de trabajo se pueden utilizar los selectores de colonias microbianas QPix?
Los selectores de colonias microbianas QPix son herramientas versátiles que se pueden integrar en diferentes flujos de trabajo. Habitualmente se utilizan en aplicaciones como cribado de clones, generación de bibliotecas de mutantes, genómica microbiana, evolución dirigida e ingeniería de cepas de alto rendimiento.
¿Cómo optimizan los selectores de colonias microbianas QPix la investigación científica?
Los selectores de colonias microbianas QPix optimizan el proceso de cribado de clones y selección de colonias al permitir a los investigadores analizar un gran número de colonias en menos tiempo. Esta optimización mejora la eficiencia de los proyectos de investigación, acelera los descubrimientos y facilita el análisis y experimentación posteriores.
¿Los selectores de colonias microbianas QPix pueden manejar diferentes tipos de placas de agar?
Sí, los selectores de colonias microbianas QPix están diseñados para manejar diferentes formatos de placas de agar, incluidas placas de Petri, placas multipocillo y otras placas personalizadas. El sistema es flexible y puede adaptarse a las necesidades específicas de los diferentes experimentos.
¿Cómo mejora la productividad de la investigación el software de los selectores de colonias microbianas QPix?
El software que se proporciona con los selectores de colonias microbianas QPix ofrece interfaces de usuario intuitivas para el diseño de experimentos, las personalización de los criterios de selección de colonias y el análisis de datos. Permite un seguimiento eficiente de las colonias, facilita la gestión de los flujos de trabajo y proporciona métricas detalladas para la toma de decisiones basadas en los datos.
Recursos más recientes
Aplicaciones destacadas
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/biofuels
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/blue-white-screening
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/dna-sequencing
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/monoclonal-antibodies-mabs
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/phage-display
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/protein-evolution
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/synthetic-biology
https://main--moleculardevices--hlxsites.hlx.page/fragments/applications/plasmid-production
Adelantos para los clientes

HISTORIA DE ÉXITO
Inscripta permite que los científicos realicen la edición digital del genoma...
¿Cómo podemos ayudarle a avanzar en su próximo gran descubrimiento?
Nuestros equipos altamente calificados están en primera línea con nuestros clientes, realizando demostraciones remotas o in situ de productos, seminarios web y más para ayudarle a resolver los difíciles desafíos de su investigación. ¿Cómo podemos ayudarle hoy?
Me gustaría...
Aplicaciones de los selectores de colonias microbianas QPix serie 400
Especificaciones y opciones de los selectores de colonias microbianas QPix serie 400
Recursos de los selectores de colonias microbianas QPix serie 400
Número de menciones*: 371
Últimas menciones: Si desea una lista completa, haga clic aquí.
*Fuente: https://scholar.google.com/