¿Qué es la edición génica?
La edición génica es una manipulación genética en la que el ADN genómico de un organismo vivo se deleciona, inserta, sustituye o modifica. La edición génica se dirige a un sitio específico para crear cortes en el ADN por medio de diversas técnicas y no siempre implica mecanismos de reparación. Consta de dos técnicas: inactivación y corrección.
La inactivación consiste en "apagar" un gen diana, y la corrección facilita la reparación del gen defectuoso mediante su escisión. La edición génica tiene un enorme potencial en numerosos campos, como el desarrollo de fármacos, cirugía génica, modelos animales, investigación y tratamiento de enfermedades, alimentación, biocombustibles, síntesis de biomateriales, etc.
Aunque recientemente se está empleando mucho CRISPR, una técnica importante de edición génica, la edición génica se estudió por primera vez a finales de los años 90. Desde la aparición de CRISPR, anteriormente una aplicación ambiciosa, la terapia génica se ha convertido en la aplicación de edición génica más solicitada. Esto se puede lograr a través de dos enfoques: la adición de genes, que se suman al material genético existente para compensar los genes defectuosos o ausente, y la edición de genes, que permite tratar las enfermedades modificando directamente el ADN relacionado con cada enfermedad.
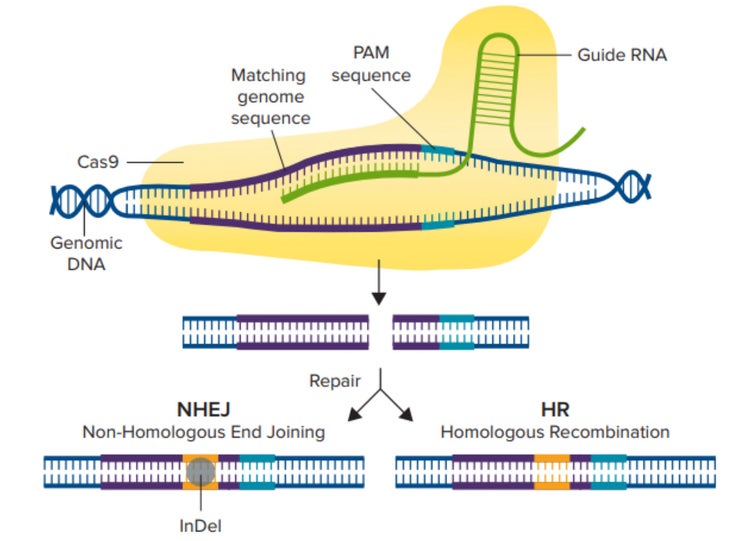
Mecanismo de CRISPR/Cas9. La enzima Cas9 se activa uniéndose primero a un ARN guía y, a continuación, a la secuencia genómica correspondiente que precede inmediatamente a la secuencia PAM de tres nucleótidos. Entonces, la enzima Cas9 genera un corte en la doble cadena y se utiliza la vía NHEJ o la HDR para reparar el ADN, lo que da lugar a una secuencia génica modificada.
Se diseña un ARN guía (ARNg) similar a un ARNcr específico de una región del gen, y la enzima Cas9 puede generar cortes en la doble cadena en la región específica del genoma de la célula huésped (figura 1). Una vez escindida la doble cadena, la célula se someterá a una de las dos siguientes vías de reparación: la vía de unión de extremos no homólogos (NHEJ) o la vía de recombinación dirigida por homología (HDR). La vía NHEJ se utiliza con frecuencia para alterar genes a través de inserciones y deleciones de bases (indels), mientras que la vía HDR se puede utilizar para introducir un gen indicador o una secuencia modificada mediante el intercambio de secuencias entre dos moléculas de ADN similares o idénticas.
Ampliación de la edición génica mediante ingeniería CRISPR
“CRISPR”: Clustered Regularly Interspaced Short Palindromic Repeats (repeticiones palindrómicas cortas regularmente interespaciadas). Estas secuencias de ADN se descubrieron por primera vez como parte del sistema inmunológico de procariotas, como bacterias y arqueas, y adquirieron importancia como herramienta de edición génica desde 2012 (Jinek et al., 2012). Esta técnica es muy prometedora para multitud de aplicaciones, como por ejemplo, en agricultura, creación de modelos de enfermedades, terapia génica o descubrimiento de fármacos, por nombrar algunos. La precisión lo convierte en una herramienta perfecta para la inserción (knock-ins), deleción (knock-outs) y demás modificaciones de las secuencias de ADN. Ha sustituido en gran medida a las tediosas y caras herramientas de edición génica existentes, como TALENS y ZFNS.
Las secuencias CRISPR contienen ADN de invasores víricos previos llamados espaciadores detrás de cada repetición palindrómica, y estas ayudan a la detección y destrucción de virus similares futuros. Entender este mecanismo (Jinek et al., 2012) permitió el uso por primera vez de CRISPR en células eucariotas (Cong, L, et al., 2013) y posteriormente en otros tipos celulares más microorganismos pertenecientes a diferentes campos. Los sistemas CRISPR-Cas9 tienen dos componentes principales que forman un complejo ribonucleoproteína. El primer componente o ARN guía se une a una secuencia de ADN complementario en el genoma y el segundo componente Cas9 de Streptococcus pyogenes (SpCas9) escinde la doble cadena en el sitio objetivo. Un motivo adyacente del protoespaciador (PAM) es donde se une inicialmente la nucleasa para realizar el corte en sentido 5'. Las diferentes nucleasas CRISPR tienen diferentes sitios PAM y una vez realizado el corte, los sistemas de reparación de las células se activan y también se inicia la edición del genoma.
Flujo de trabajo de edición génica
El flujo de trabajo de edición génica usando mecanismos de CRISPR para obtener una línea celular modificada confirmada consta de varios pasos. La optimización efectiva de estos pasos, usando las herramientas correctas contribuye a un proceso eficaz para acortar tiempo, esfuerzos y costes de diversos avances científicos. Esta estrategia ayuda a acelerar la I+D, revoluciona el descubrimiento de fármacos, la cura de enfermedades, la producción de cultivos modificados genéticamente, etc. Discutimos los pasos involucrados y las soluciones efectivas que ofrecemos para ayudar a las comunidades científicas de todo el mundo a lograr sus objetivos a través de la edición génica.
Soluciones de investigación para validar ediciones génicas CRISPR/Cas9
La familia de instrumentos de Molecular Devices puede utilizarse de forma eficaz para realizar o seleccionar experimentos, lo que garantiza el éxito de los esfuerzos de edición génica. El nuevo generador de imágenes CloneSelect Imager Florescence (CSI-FL) proporciona garantía de monoclonalidad desde el día 0 tras la impresión de células individuales, eficiencia de la transfección, confluencia celular y datos de cribado con fluorescencia multicanal para validar la eficacia de la edición génica con tiempos de seguimiento más cortos, bajo riesgo de exceso de pases y robótica.
Además, se puede utilizar nuestro lector de microplacas multimodo SpectraMax i3x para evaluar la eficiencia de la transfección, monitorizar el crecimiento celular, cuantificar el ADN y las proteínas y validar las ediciones CRISPR/Cas9 a través del análisis mediante inmunotransferencia ScanLater. Se pueden adquirir imágenes de alta calidad de autofagosomas utilizando el sistema ImageXpress Micro Confocal, mientras que el software MetaXpress HCI puede identificar y cuantificar autofagosomas individuales de cada célula, lo que nos permite analizar los cambios fenotípicos que tienen lugar en la edición génica CRISPR/Cas9.
